El cólera continúa representando uno de los mayores desafíos para la salud pública mundial, con entre 1,3 y 4 millones de casos estimados cada año y decenas de miles de muertes reportadas globalmente. Esta enfermedad, provocada por la bacteria Vibrio cholerae, se transmite principalmente por el consumo de agua y alimentos contaminados, y afecta de manera desproporcionada a poblaciones en regiones con acceso limitado a saneamiento seguro y agua potable.
Factores como los conflictos armados, el cambio climático y los desplazamientos poblacionales han impulsado un resurgimiento notable de brotes epidémicos en los últimos años. En respuesta a esta situación alarmante —que en 2023 afectaba ya a 43 países y mostraba una elevada mortalidad, especialmente entre niños—, la Organización Mundial de la Salud (OMS) clasificó el cólera como una emergencia de grado 3, su nivel más alto de alerta interna.
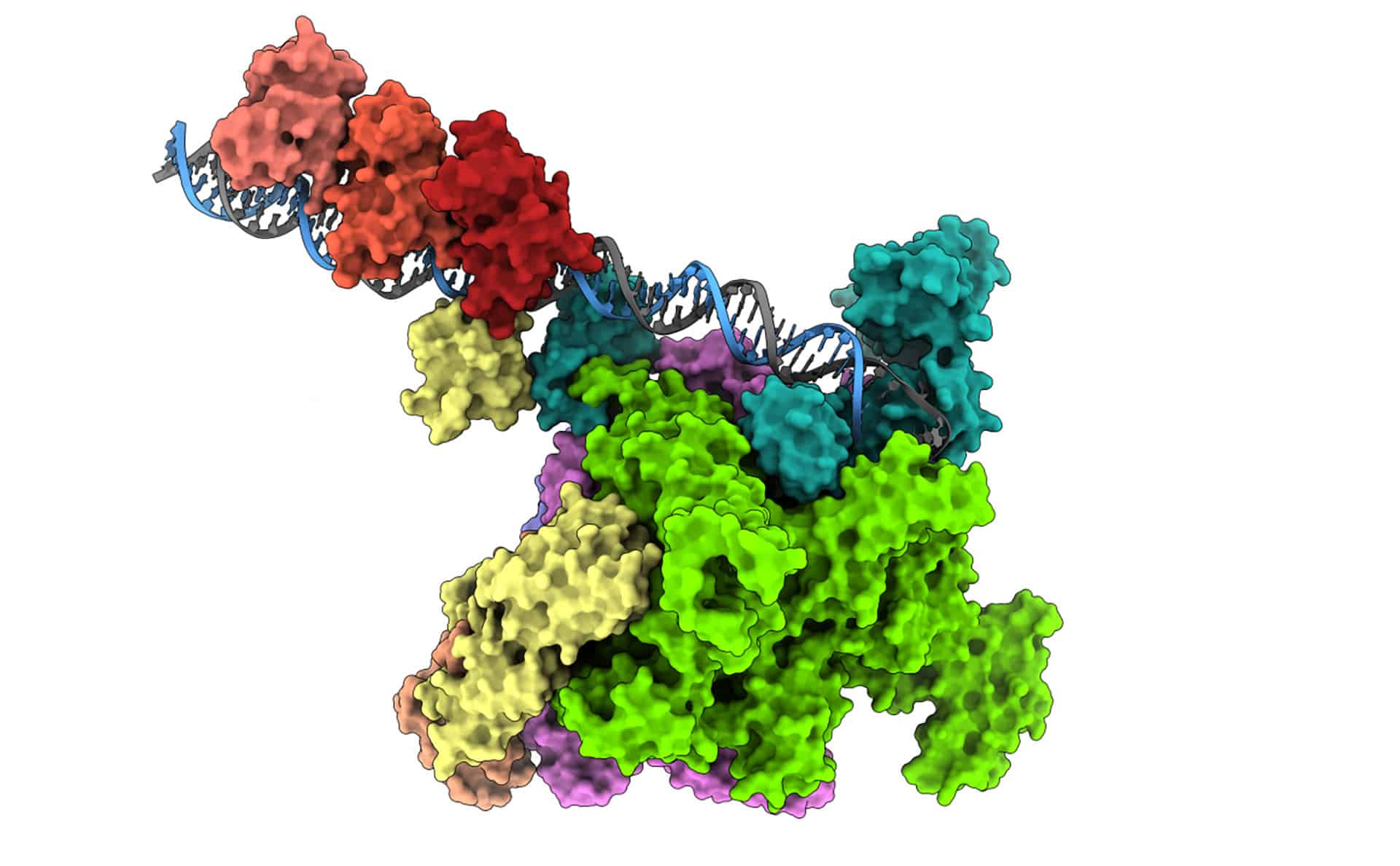
En este contexto de expansión global de la enfermedad, un avance científico reciente arroja luz sobre los mecanismos moleculares que permiten a la bacteria volverse letal. Un estudio internacional, publicado en Science Advances, ha revelado por primera vez la arquitectura molecular detallada de cómo dos factores de transcripción clave —ToxR y TcpP— activan la virulencia de Vibrio cholerae en el intestino humano.
Estos factores detectan señales del entorno intestinal, como la presencia de sales biliares y bajos niveles de oxígeno, y se unen al ADN bacteriano para iniciar una cascada regulatoria. Esta cascada culmina en la producción de la toxina del cólera (responsable de la diarrea acuosa masiva) y del pilus corregulado por toxinas (TCP), una estructura filamentosa que permite a la bacteria adherirse firmemente a las paredes del intestino delgado.
Aunque ToxR y TcpP fueron identificados hace décadas como reguladores principales de la infección, hasta ahora se desconocía con precisión cómo interactuaban con la ARN polimerasa (RNAP), el motor celular que transcribe el ADN a ARN. Utilizando criomicroscopía electrónica, el equipo internacional —integrado por investigadores del Instituto de Biología Molecular de Barcelona (IBMB-CSIC), el IRB Barcelona, el Laboratorio Europeo de Biología Molecular (EMBL) en Heidelberg (Alemania) y la Universidad de Detroit Mercy (EE.UU.)— ha resuelto esta estructura en 3D.
Contrario a lo esperado, el mecanismo no implica un cambio conformacional drástico en la polimerasa, como ocurre en muchos otros reguladores bacterianos. En cambio, ToxR y TcpP funcionan como anclas moleculares que estabilizan una región específica de la enzima (el dominio alfa-CTD) directamente sobre el ADN promotor, fijándola en una configuración productiva para la transcripción de genes de virulencia.